日常工作之读文档,了解参数改变、新方法功能,对已有流程进行修改。
各种文档看一边遍一遍,使用需要又一次看 SnpEff (http://snpeff.sourceforge.net/index.html ),进行一些整理如下,不过还是建议原文,鬼知道我会在哪里写错理解错。
运行
annotation
1
2
3
4
5
6
7
8java -jar snpEff.jar eff genome input.vcf >output.vcf
-hgvs, 默认开启,使用 HGVS 变异描述语法对蛋白替换进行描述
-fi, 提供bed文件,只对bed区域内位点进行注释
-no-downstream, -no-intergenic, -no-intron, -no-upstream, -no-utr, -no [effectType], 参数还可对根据注释类型对注释进行初过滤
-canon, 只使用基因对应最长转录本(作为权威转录本)进行注释;参数-canonList可以自行提供基因权威转录本,格式为“GeneID transcript_ID”
-noStats, 不输出注释统计信息
-v, 话痨模式,输出各种运行信息
database
人、小鼠等物种都有已经 build 的数据库,可以直接下载,链接:https://sourceforge.net/projects/snpeff/files/databases/v4_3/,注释选择基因组版本。
1
2
3
4java -jar snpEff.jar databases # 查看可用数据库
java -jar snpEff.jar download -v GRCh37.75 # 下载数据库 GRCh37.75
Q, 使用执行程序下载数据库时容易断,一般建议手动下载,或者wget等其他方式软件还提供了自行构建数据库,详细用法如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19添加配置信息
echo 'GRCh37.70.genome : Human ' >>~/snpEff/snpEff.config
Create directoy for this new genome
mkdir -p ~/snpEff/data/GRCh37.70 && cd ~/snpEff/data/GRCh37.70
Get annotation files
wget -c -O genes.gtf.gz ftp://ftp.ensembl.org/pub/release-70/gtf/homo_sapiens/Homo_sapiens.GRCh37.70.gtf.gz
Get the genome
wget -c -O sequences.fa.gz ftp://ftp.ensembl.org/pub/release-70/fasta/homo_sapiens/dna/Homo_sapiens.GRCh37.70.dna.toplevel.fa.gz
Optional, Download CDSs
wget -c -O cds.fa.gz ftp://ftp.ensembl.org/pub/release-70/fasta/homo_sapiens/cdna/Homo_sapiens.GRCh37.70.cdna.all.fa.gz
Optional, Download proteins
wget -c -O protein.fa.gz ftp://ftp.ensembl.org/pub/release-70/fasta/homo_sapiens/pep/Homo_sapiens.GRCh37.70.pep.all.fa.gz
Optional, Download regulatory annotations
wget -c -O regulation.gff.gz ftp://ftp.ensembl.org/pub/release-70/regulation/homo_sapiens/AnnotatedFeatures.gff.gz
Building a database from GFF files
cd ~/snpEff && java -jar snpEff.jar build -gff3 -v GRCh37.70
rm -r ~/snpEff/data/GRCh37.70此外,snpEff 构建数据库还支持 RefSeq, GenBank 等多种数据输入格式,具体用法查看原文档。
注意物种 codon 选择,codon 可以在 https://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi 中查看,codon可以在
~/snpEff/snpEff.config文件中设置:1
2
3
4# 设置特异 codon
codon.Invertebrate_Mitochondrial: TTT/F, TTC/F, TAC/Y, TAA/*, ATG/M+, ATG/M+, ACT/T, ...
# 物种选择恰当的 codontable
dm3.M.codonTable : Invertebrate_Mitochondrial # the chromosome 'M' from fly genome (dm3) uses Invertebrate_Mitochondrial codon table
dump
从数据库中提取出注释信息
1
2
3java -jar snpEff.jar dump -v -bed GRCh37.70 > GRCh37.70.bed
[-bed |-txt ] 设置数据格式
[-0 |-1 ] 设置坐标系统,0-based 或者 1-based
注释结果
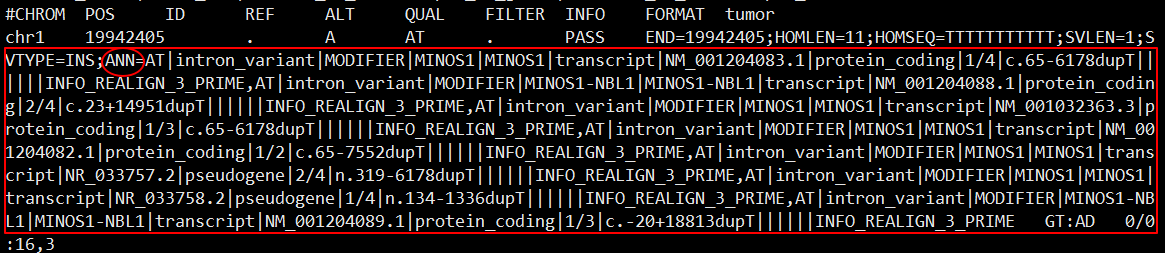
可以看到注释信息被添加到了 VCF 中每个变异 INFO 信息中,以 ANN= 特征开始,详细的注释说明可查看官方文档: http://snpeff.sourceforge.net/VCFannotationformat_v1.0.pdf。因为基因多个转录本、相互重叠基因等原因,可以看到变异位点被多次注释,需根据实际需求进行筛选。